Es gibt eine große Auswahl an Linux-Bioinformatik-Tools, die in diesem Bereich seit langem weit verbreitet sind. Die Bioinformatik wurde in vielerlei Hinsicht charakterisiert; Es wird jedoch häufig als eine Kombination aus Mathematik, Berechnung und Statistik zur Analyse biologischer Informationen definiert. Das Hauptziel des Bioinformatik-Tools ist es, einen effizienten Algorithmus zu entwickeln, damit Sequenzähnlichkeiten entsprechend gemessen werden können.
Beste Bioinformatik-Tools für Linux
Dieser Artikel wurde geschrieben, indem er sich auf die Bioinformatik-Tools konzentriert, die auf der Linux-Plattform verfügbar sind. Alle effizienten Tools wurden ausführlich besprochen und überprüft. Darüber hinaus finden Sie in diesem Artikel die wesentlichen Funktionen, Eigenschaften und Download-Links. Gehen wir es also durch.
1. geWorkbench
geWorkbench kann mit genome workbench ausgearbeitet werden ist ein Java-basiertes Bioinformatik-Tool, das für integrierte Genomik arbeitet. Seine Komponentenarchitekturen ermöglichen speziell entwickelte Plug-Ins, die in komplizierte Bioinformatik-Anwendungen konfiguriert werden können. Derzeit sind mehr als 70 Plug-ins zur Unterstützung, Visualisierung und Analyse von Sequenzdaten verfügbar.
Funktionen von geWorkbench
- Es ist in vielen computergestützten Analysewerkzeugen enthalten, nämlich t-Test, selbstorganisierende Karten und hierarchisches Clustering und so weiter.
- Es ist mit molekularen Interaktionsnetzwerken, Proteinstruktur und Proteindaten ausgestattet.
- Es bietet Genintegrations- und Annotationspfade und sammelt Daten aus kuratierten Quellen für die Genontologie-Anreicherungsanalyse.
- In diesem Tool werden Komponenten mit der Plattformverwaltung von Ein- und Ausgängen integriert.
2. BioPerl
BioPerl ist eine Sammlung von Perl-Werkzeugen, die in der Linux-Plattform als Bioinformatik-Werkzeug für Computer-Molekularbiologie weit verbreitet sind. Es wird in den Bereichen der Bioinformatik kontinuierlich in einer Reihe von Standard-CPAN-Stilen verwendet. Dieses Linux-Bioinformatik-Tool ist gut dokumentiert und in Perl-Modulen frei verfügbar. Da diese Module objektorientiert sind, sind sie voneinander abhängig, um die Aufgabe zu erfüllen.
Merkmale von BioPerl
- Aus den lokalen und isolierten Datenbanken greift dieses Bioinformatik-Tool auf Nukleotid- und Peptidsequenzdaten zu.
- Es manipuliert verschiedene Sequenzen und transformiert auch die Form von Datenbank- und Dateiaufzeichnungen.
- Es fungiert als Bioinformatik-Suchmaschine, bei der es nach ähnlichen Sequenzen, Genen und anderen Strukturen auf genomischer DNA sucht.
- Durch Generieren und Manipulieren von Sequenzalignments werden maschinenlesbare Sequenzannotationen entwickelt.
3. UGENE
UGENE ist eine kostenlose Open Source und eine Reihe von integrierenden Bioinformatik-Tools für Linux. Die gemeinsame Benutzeroberfläche ist in die meistgenutzten und vertrauten Bioinformatik-Anwendungen integriert. Zahlreiche biologische Datenformate sind mit seinen Toolkits kompatibel; somit können Daten von entfernten Quellen abgerufen werden. Dieses Bioinformatik-Tool verwendet Multicore-CPUs und -GPUs, um die maximal mögliche Leistung zur Optimierung seiner Rechenaktivitäten bereitzustellen.
Funktionen von UGENE
- Die grafische Benutzeroberfläche bietet mehrere Funktionen, zum Beispiel Chromatogramm-Visualisierung, Multiple-Align-Editor und visuelle und interaktive Genome.
- Es ebnet den Weg für eine 3D-Ansicht im PDB- und MMDB-Format zusammen mit der Unterstützung des Anaglyph-Stereomodus.
- Es erleichtert die phylogenetische Baumansicht, die Dot-Plot-Visualisierung und der Abfrage-Designer kann nach komplizierten Anmerkungsmustern suchen.
- Es kann den Weg für benutzerdefinierte rechnerische Arbeitsabläufe für den Arbeitsablauf-Designer ebnen.
4. Biojava
Biojava ist Open Source und ausschließlich für das Projekt konzipiert, um die erforderlichen Java-Tools zur Verarbeitung biologischer Daten bereitzustellen. Es funktioniert für weite Bereiche von Datensätzen, zum Beispiel analytische und statistische Routinen, Parser für gängige Dateiformate. Darüber hinaus erleichtert es die Manipulation von Sequenzen und 3D-Strukturen. Dieses Bioinformatik-Tool für Linux zielt darauf ab, die schnelle Anwendungsentwicklung für biologische Datensätze zu beschleunigen.
Merkmale von Biojava
- Es enthält Klassendateien und Objekte und ist ein Paket, das Java-Code für eine Vielzahl von Datensätzen implementiert.
- Biojava kann in verschiedenen Projekten wie Dazzel, Bioclips, Bioweka und Genious verwendet werden, die für verschiedene Zwecke verwendet werden.
- Es funktioniert für Dateiparser zusammen mit DAS-Clients und Serverunterstützung.
- Es wird zur Durchführung von Sequenzanalysen für GUIs verwendet und kann auf BioSQL- und Ensembl-Datenbanken zugreifen.
5. Biopython
Das Bioinformatik-Tool Biophython, das von einem internationalen Entwicklerteam entwickelt und im Python-Programm geschrieben wurde, wird für biologische Berechnungen verwendet. Es bietet Zugriff auf eine große Auswahl an Bioinformatik-Dateiformaten, nämlich BLAST, Clustalw, FASTA, Genbank, und ermöglicht den Zugriff auf Online-Dienste wie NCBI und Expasy.

Merkmale von Biopython
- Es wird mit Python-Modulen angesammelt, die daran arbeiten, eine Sequenz mit interaktiver und integrierter Natur zu erstellen.
- Dieses Bioinformatik-Tool kann in verschiedenen Sequenzen ausgeführt werden, z. B. Übersetzung, Transkription und Gewichtsberechnungen.
- Dieses Tool ist ausschließlich angereichert; somit werden Proteinstruktur und Sequenzformat effizient verwaltet.
- Dieses Linux-Bioinformatik-Tool funktioniert für Alignments; somit kann ein Standard zur Erstellung und Handhabung von Substitutionsmatrizen etabliert werden.
6. InterMine
InterMine ist ein Open-Source-Bioinformatik-Tool für Linux, das als Data Warehouse zur Integration und Analyse biologischer Daten fungiert. Als Software können Nutzer diese auf ihrem Gerät installieren und Daten auf der Webseite zur Verfügung stellen. Es wird angenommen, dass es sich um eine der dynamischsten Datentabellen handelt, die Daten leicht aufschlüsseln können, und es erleichtert das Filtern von Daten. Was ist eine zusätzliche Spalte, um zur Berichtsseite zu navigieren?
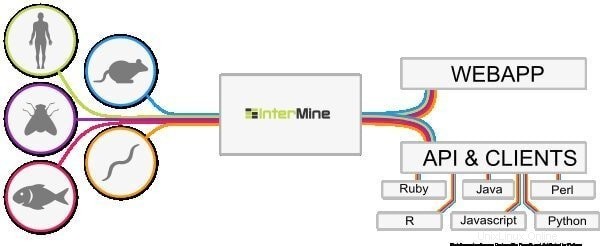
Funktionen von InterMine
- Es funktioniert mit einem einzelnen Objekt, z. B. einem Gen, Protein oder einer Bindungsstelle, und mehreren Listen, z. B. einer Liste von Genen oder einem Listenprotein.
- Es kann in mehreren Sprachen betrieben werden; somit können verschiedene Abfragen zu biometrischen Informationen in mehreren Sprachen durchsucht werden.
- In dieser Software sind vier Suchwerkzeuge verfügbar:Vorlagensuche, Schlüsselwortsuche, Abfrageerstellung und Regionssuche.
- Es unterstützt verschiedene Formate wie Chado, GFF3, FASTA, GO &Gene Association Files, UniProt XML, PSI XML, In Paranoid Orthologe und Ensembl.
7. IGV
IGV, das als interaktiver Genomik-Viewer entwickelt wurde, gilt als eines der effektivsten Visualisierungstools, das problemlos auf eine umfangreiche und interaktive Genomik-Datenbank zugreifen kann. Es kann eine Vielzahl von Datentypen mit genomischer Annotation zusammen mit Array-basierten und Sequenzdaten der nächsten Generation anbieten. Genau wie Google Maps kann es durch einen Datensatz navigieren und das nahtlose Zoomen und Schwenken über das Genom erleichtern.
Funktionen von IGV
- Es bietet eine flexible Integration von weiten Bereichen genomischer Datensätze, einschließlich ausgerichteter Sequenz-Reads, Mutationen, Kopienzahlen und so weiter.
- Durch die Verwendung von effizienten Dateiformaten mit mehreren Auflösungen beschleunigt es die Erforschung des massiven unterstützenden Datensatzes in Echtzeit.
- Unter Hunderten und teilweise bis zu Tausenden von Samples ermöglicht es die gleichzeitige Visualisierung verschiedener Datentypen.
- Es ermöglicht das Laden von Datensätzen aus lokalen und entfernten Quellen, einschließlich Cloud-Datenquellen, um eigene und öffentlich verfügbare genomische Datensätze zu beobachten.
8. GROMACS
GROMACS ist ein dynamischer Molekularsimulator, der in Analyse- und Erstellungstools enthalten ist. Es ist ein Paket mit Vielseitigkeit und beabsichtigt, an der Molekulardynamik zu arbeiten; Beispielsweise kann es die Newtonsche Bewegungsgleichung von Hunderten bis Tausenden von Teilchen simulieren. Es wurde so programmiert, dass es auf biochemischen Molekülen in früheren Stadien, nämlich Proteinen und Lipiden, die durch komplizierte Wechselwirkungen verbunden sind, funktioniert.
Funktionen von GROMACS
- Dieses Linux-Informatiktool ist benutzerfreundlich, enthält Topologien und Parameterdateien und ist in Klartext geschrieben.
- Skriptsprache wurde nicht verwendet; Daher werden alle Programme mit einer einfachen Befehlszeilenoption für Eingabe- und Ausgabedateien bedient.
- Wenn etwas schief geht, werden viele Fehlermeldungen und Konsistenzprüfungen durchgeführt.
- Alle Programme werden durch die integrierte grafische Benutzeroberfläche erleichtert.
9. Taverna Werkbank
Die Taverna Workbench ist ein Open-Source-Tool, das zum Entwerfen und Ausführen von Bioinformatik-Workflows programmiert ist, die vom myGrid-Projekt erstellt wurden. In dieses Tool kann eine Reihe von Software integriert werden, darunter SOAP- und REST-Webservice. Es arbeitet mit verschiedenen Organisationen wie dem European Bioinformatics Institute, der DNA Databank of Japan, dem National Center for Biotechnology Information, SoapLab, BioMOBY und EMBOSS zusammen.
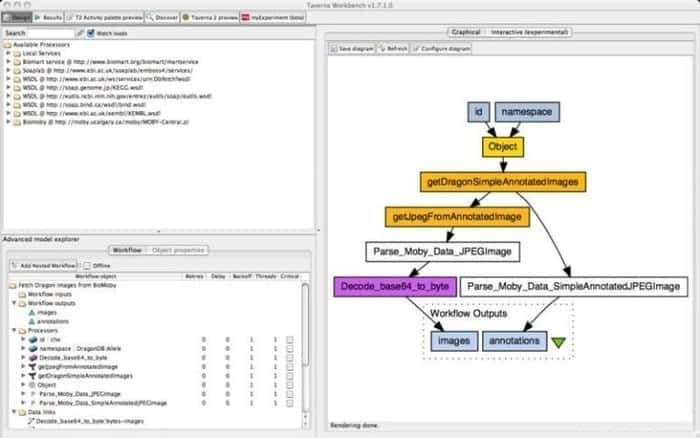
Funktionen von Taverna Workbench
- Es ist vollständig auf den grafischen Workflow zum Finden, Entwickeln und Ausführen von Workflows ausgelegt.
- Es wurde mit einem vollständig grafischen Arbeitsablauf entwickelt; außerdem werden diskrete tabs für die gestaltung verwendet.
- Anmerkungen dienen zur Beschreibung von Arbeitsabläufen, Diensten, Eingaben und Ausgaben mit einer integrierten Hilfefunktion.
- Zuvor verwendeter Workflow wird in diesem Tool gespeichert, auch wenn es in der Datei verwendete Workflow-Eingaben speichern kann.
10. PRÄGUNG
EMBOSS, das impliziert die European Molecular Biology Open Software Suite. Es ist ein Softwarepaket, das für die Bedürfnisse der Molekularbiologie-Community entwickelt wurde. Dieses Linux-Bioinformatik-Tool kann für verschiedene Zwecke verwendet werden. Beispielsweise funktioniert es automatisch in verschiedenen Datenformaten. Darüber hinaus kann es Daten sequenziell von der Webseite sammeln.
Funktionen von EMBOSS
- EMBOSS ist in Hunderten von Anwendungen enthalten, nämlich Sequenzabgleich und schnelle Datenbanksuche mit Sequenzmustern.
- Zusätzlich verfügt es über eine Proteinmotividentifikation, einschließlich Domänenanalyse und Nukleotidsequenzmusteranalyse.
- Sein Toolkit wurde in geeigneter Weise entwickelt, um die Anwendung und den Arbeitsablauf der Bioinformatik zu adressieren.
- Es wurde mit zusätzlichen Bibliotheken programmiert, um auch viele andere relevante Probleme zu behandeln.
11. Clustal Omega
Clustal Omega arbeitet an Proteinen, und RNA/DNA ist ein multiples Sequenz-Alignment-Programm, das für allgemeine Zwecke entwickelt wurde. Es kann Millionen von Datensätzen in angemessener Zeit effizient verarbeiten; Darüber hinaus produziert es hochwertige MSAs. In diesem Linux-Bioinformatik-Tool gibt es einen Prozess, bei dem der Benutzer die Dateisequenz im Standardmodus belassen muss. Das wird ausgerichtet und gruppiert, um einen Leitbaum zu erzeugen, und das ermöglicht letztendlich die Bildung einer progressiven Ausrichtungssequenz.
Merkmale von Clustal Omega
- Es erleichtert das Angleichen vorhandener Alignments aneinander und darüber hinaus das Angleichen einer Sequenz an ein Alignment zur Verwendung eines Hidden-Markov-Modells.
- Es gibt eine Funktion namens externes Profil-Alignment, die sich auf eine neue Sequenz von Homologen für das Hidden-Markov-Modell bezieht.
- HMMs werden für den Clustal Omega für die Alignment-Engine aus dem HHaign-Paket von Johannes Soeding verwendet.
- Clustal Omega erlaubt drei Arten von Sequenzeingaben:das Profil, die Sequenz ausrichten und HMM.
12. BLAST
Das Basic Local Alignment Search Tool oder BLAST wird verwendet, um die Ähnlichkeit zwischen biologischen Sequenzen zu finden. Es kann relevante Übereinstimmungen zwischen Nukleotid- und Proteinsequenzen finden und deren statistische Bedeutung zeigen. Abfragesequenzen werden mit verschiedenen Arten von BLAST strukturiert. Darüber hinaus wird dieses Tool weitgehend mit unbekannten Genen in verschiedenen Tieren kultiviert und ermöglicht die Kartierung sequenzbasierter Datensätze durch qualitative Analyse.

Funktionen von BLAST
- Das megaBLAST Nukleotid-Nukleotid bietet die Möglichkeit, nach sehr ähnlichen Arten von Sequenzen zu suchen und diese zu optimieren.
- Außerdem funktioniert das BLASTN-Nukleotid-Nukleotid etwas anders, da es nach Distanzsequenzen sucht.
- Darüber hinaus führt BLASTP das Auffinden von Protein-Protein-Beziehungen und -Vergleichen durch, und seine Formel wird für verschiedene andere Forschungszwecke verwendet.
- TBLASTN konzentriert sich auf die Nukleotidabfrage gegen den Proteindatensatz und kann die Datenbank im Handumdrehen übersetzen.
13. Bettzeug
Die Bioinformatik-Software von Bedtool ist ein Schweizer Taschenmesser für weite Bereiche der Genomanalyse. Die genomische Arithmetik verwendet dieses Werkzeug sehr häufig, was impliziert, dass sie damit die Mengenlehre finden kann. Bedtools erleichtern zum Beispiel das Zählen, Ergänzen und Mischen von Schnittmengen, das Zusammenführen genomischer Intervalle aus mehreren Dateien und das Generieren eines bestimmten Genomformats wie BAM, BED, GFF/GTF, VCF.
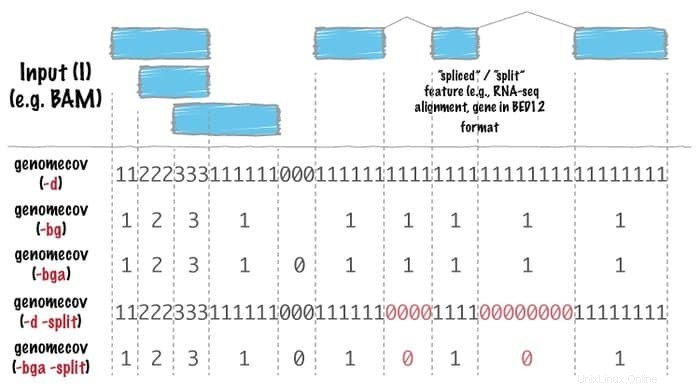
Funktionen von Bedtools
- In diesem Linux-Bioinformatik-Tool ist jedes so konzipiert, dass es eine besonders einfache Aufgabe ausführt, z. B. zwei Intervalldateien schneidet.
- Die komplizierte und ausgefeilte Analyse wird durch die Verwendung einer Kombination von Bedtools durchgeführt.
- Dieses Tool wurde im Quinlan-Labor der Utah University von einem Forscher der Gruppe entwickelt.
- Da es viele Optionen in diesem Tool gibt, kann es für viele Zwecke im Bereich der Bioinformatik verwendet werden.
14. Bioclipse
Bioclipse Linux-Bioinformatik-Tool, das mit Workbench for Life Science definiert ist, ist eine Java-basierte Open-Source-Software. Es funktioniert auf der visuellen Plattform, die die Chemo- und Bioinformatik-Eclipse-Rich-Client-Plattform umfasst. Es ist mit einer Plugin-Architektur ausgestattet. Das impliziert die hochmoderne Plugin-Architektur, außerdem Funktionalität und visuelle Schnittstellen von Eclipse, wie z. B. Hilfesystem, Software-Updates sind ebenfalls enthalten.
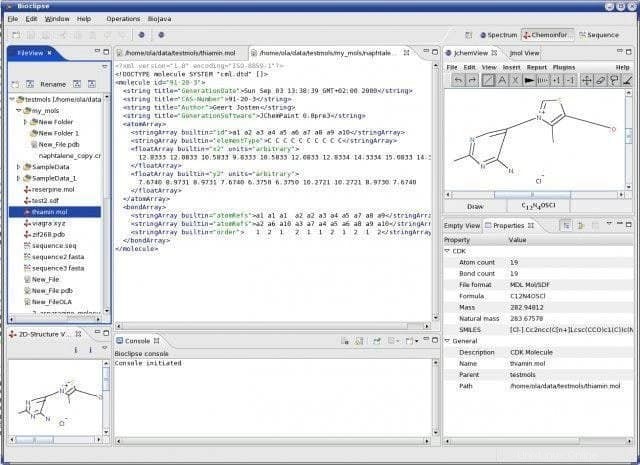
Funktionen von Bioclipse
- Biologische Sequenzen, nämlich RNA, DNA und Protein, werden mit dem Bioclipse verwaltet.
- Biojava hilft auch bei der Bereitstellung grundlegender Bioinformatik-Funktionalität; auch grafische Editoren für Sequenzalignments.
- Es wird für die Pharmakologie und Arzneimittelentdeckung zusammen mit der Entdeckung des Metabolismus verwendet.
- Schließlich arbeitet es an der Funktionalität des semantischen Webs, beim Durchsuchen umfangreicher Sammlungen von Verbindungen und beim Bearbeiten chemischer Strukturen.
15. Bioleiter
Die in der Linux-Plattform weit verbreitete Bioinformatik ist ein quelloffenes und kostenloses Bioinformatik-Tool, das in der medizinischen Biologie kohärent für Hochdurchsatzanalysen verwendet wird. Es verwendet hauptsächlich statistische R-Programmierung; es enthält jedoch auch eine andere Programmiersprache. Diese Software wurde entwickelt, indem sie sich auf einige Ziele konzentriert; Beispielsweise zielt es darauf ab, eine kollaborative Entwicklung zu etablieren und sicherzustellen, dass innovative Software immens genutzt wird.
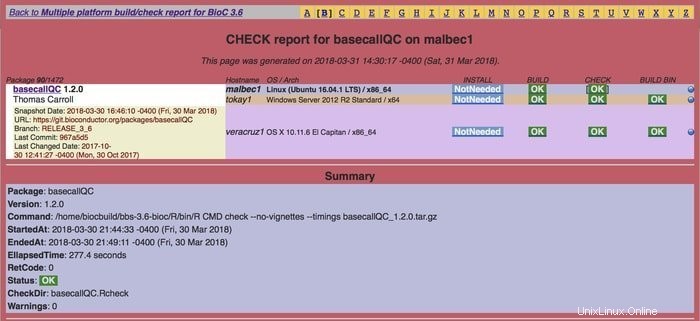
Merkmale von Bioconductor
- Diese Software kann eine Reihe von Daten analysieren, z. B. Oligonukleotid-Arrays, Sequenzanalysen, Durchflusszytometer, und eine robuste grafische und statistische Datenbank erstellen.
- Das Vorhandensein von Vignetten und Dokumenten in jedem Binocular-Paket kann eine textuelle und aufgabenorientierte Beschreibung der Funktionalität dieses Pakets bieten.
- Es kann Echtzeitdaten zum zugehörigen Mikroarray und andere genomische Daten zusammen mit biologischen Metadaten generieren.
- Außerdem kann es Express-Gene wie LIMMA, cDNA-Arrays, Affy-Arrays, RankProd, SAM, R/maanova, Digital Gene Expression usw. analysieren.
16. AMPHORA
AMPHORA, das für Automated Phylogenomic infeRence Application steht, ist ein Open-Source-Bioinformatik-Workflow-Tool. Eine andere Version von AMPHORA, die AMPHORA2 genannt wird, hat bakterielle und 104 archaeale phylogenetische Markergene. Noch wichtiger ist, dass es Informationen zwischen phylogenetischen und metgenetischen Datensätzen erstellt.
Merkmale von AMPHORA
- Da es sich um einzelne Gene handelt, ist AMPHORA2 am besten geeignet, um die taxonomische Zusammensetzung von Bakterien abzuleiten.
- Darüber hinaus kann es auch die taxonomische Zusammensetzung archaealer Gemeinschaften aus der metagenomischen Schrotflintensequenz ableiten.
- Am Anfang wurde AMPHORA verwendet, um die metagenomischen Daten der Sargassosee zu analysieren.
- Heutzutage wird AMPHORA2 jedoch zunehmend verwendet, um relevante metagenomische Daten in dieser Hinsicht zu analysieren.
17. Anduril
Anduril ist eine auf Open-Source-Komponenten basierende Bioinformatik-Software für Linux, die zur Erstellung eines Workflow-Frameworks für die wissenschaftliche Datenanalyse dient. Dieses Tool wurde vom Systems Biology Laboratory der Universität Helsinki entwickelt. Dieses Bioinformatik-Tool für Linux wurde entwickelt, um eine effiziente, flexible und systematische Datenanalyse zu ermöglichen, insbesondere im Bereich der biomedizinischen Forschung.
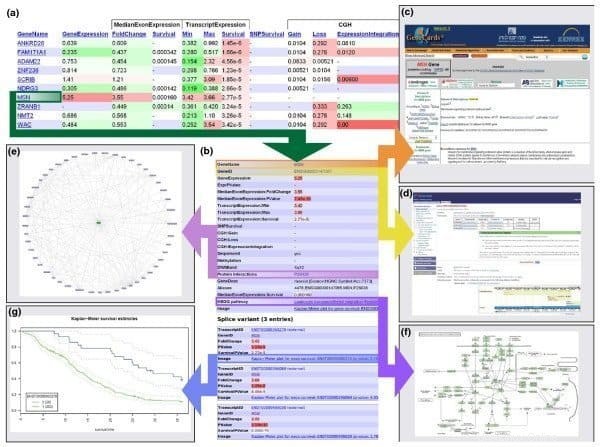
Eigenschaften von Abduril
- Es funktioniert in einem Arbeitsablauf, in dem verschiedene Verarbeitungssysteme miteinander verbunden sind; zum Beispiel; eine Ausgabe eines Prozesses kann als Eingabe für andere dienen.
- Das primäre Anduril-Tool ist in Java geschrieben, während andere Komponenten in anderen Anwendungen geschrieben sind.
- In seinen verschiedenen Schritten finden zahlreiche Aktivitäten statt, wie z. es erstellt Daten, generiert Berichte und importiert auch Daten.
- Die Workflow-Konfiguration kann mit einer einfachen, leistungsstarken Skriptsprache, nämlich Andurilscript, vorgenommen werden.
18. LabKey-Server
LabKey Server ist eine bevorzugte Wahl für Wissenschaftler, die in den Labors eingesetzt werden, um biomedizinische Daten zu integrieren, zu analysieren und zu teilen. In diesem Tool wird ein sicheres Datenrepository verwendet, das webbasierte Abfragen, Berichte und Zusammenarbeit in einer Vielzahl von Datenbanken erleichtert. Zusammen mit der gegebenen zugrunde liegenden Plattform können viele weitere wissenschaftliche Instrumente in dieser Anwendung hinzugefügt werden.
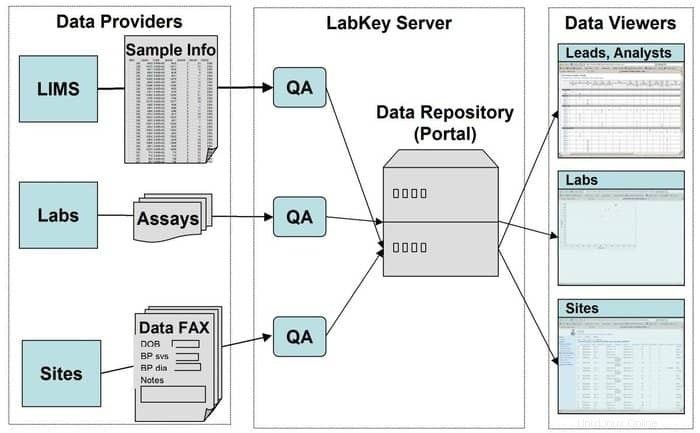
Funktionen von LabKey Server
- LabKey Server ist mit allen Arten von biomedizinischen Daten ausgestattet. Zum Beispiel Durchflusszytometrie, Mikroarray, Massenspektrometrie, Mikroplatte, ELISpot, ELISA usw.
- In diesem Tool führt eine anpassbare Datenverarbeitungspipeline alle relevanten Aktivitäten aus.
- Es ist mit Beobachtungsstudien ausgestattet, die das Management von langfristigen, groß angelegten Studien von Teilnehmern unterstützen.
- Proteomik wird zur Verarbeitung von Hochdurchsatz-Massenspektrometriedaten mit einem speziellen Tool, nämlich X! Tandem.
19. Mothur
Mothur ist ein Open-Source-Bioinformatik-Tool, das im biomedizinischen Bereich zur Verarbeitung biologischer Daten weit verbreitet ist. Es ist ein Softwarepaket, das häufig zur Analyse von DNA aus unkultivierten Mikroben verwendet wird. Mothur ist ein Linux-Bioinformatik-Tool, das Daten verarbeiten kann, die aus DNA-Sequenzierungsmethoden generiert wurden, einschließlich 454-Pyro-Sequenzierung.
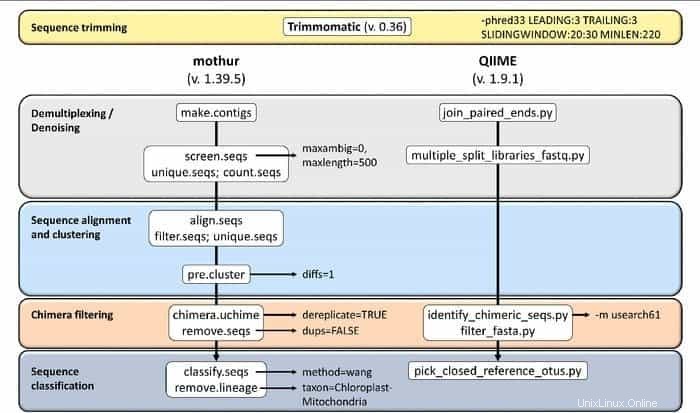
Merkmale von Mothur
- Es handelt sich um eine Einzelpaket-Software, die in der Lage ist, Community-Daten zu analysieren und eine Sequenz zu erstellen.
- Mit diesem Tool wird umfangreiche Community-Dokumentationsunterstützung und eine andere Form der Unterstützung bereitgestellt.
- Es wird angenommen, dass Mothur das bekannteste Bioinformatik-Tool zur Analyse von 16S-rRNA-Gensequenzen ist.
- Eine spezielle Community und Tutorials sind in diesem Tool verfügbar, um zu informieren, wie man Sanger, PacBio, IonTorrent, 454 und Illumina (MiSeq/HiSeq) verwendet.
20. VOTCA
VOTCA steht für Versatile Object-Oriented Toolkit for Coarse-Graining Applications, das als effizientes Bioinformatik-Tool mit einem Coarse-Grained-Modellierungspaket gebrandmarkt wird, das hauptsächlich molekularbiologische Daten analysiert. Es zielt darauf ab, systematische Grobkörnungstechniken zusammen mit der Simulation mikroskopischer Ladung zu entwickeln, um ungeordnete Halbleiter zu transportieren.
Funktionen von VOTCA
- VOTCA besteht hauptsächlich aus drei Hauptteilen:dem Coarse-graining Toolkit, dem Charge Transport Toolkit und dem Excitation Transport Toolkit.
- Alle drei Kernfunktionen stammen aus der VOTCA-Toolbibliothek, die gemeinsam genutzte Prozeduren implementiert.
- VOTCA verwendet grobkörnige Methoden, um die besten Ergebnisse aus relevanten Aktivitäten zu erzielen.
- Diese Software ist mit einem Erregungstransport-Toolkit ausgestattet, von dem Orca-DFT-Pakete in erheblichem Umfang unterstützt werden.
Abschließender Gedanke
Um das Ganze zusammenzufassen, sei hier erwähnt, dass alle oben genannten Anwendungen der Bioinformatik in diesem Bereich umfassend genutzt werden. Diese Linux-Bioinformatik-Tools werden seit langem in der Medizin, Pharmakologie, Arzneimittelerfindung und in relevanten Bereichen eingesetzt. Schließlich werden Sie gebeten, Ihre zwei Pfennige für diesen Artikel zu hinterlassen. Wenn Sie diesen Artikel interessant finden, vergessen Sie bitte nicht, ihn zu liken, zu teilen und zu kommentieren. Wir freuen uns über Ihren wertvollen Kommentar.